在探索生命科学的奥秘中,紫杉醇这一神奇的药物始终占据着举足轻重的地位。作为一种抗击癌症的利器,紫杉醇的结构复杂而独特,它由一个高度活跃的碳骨架和一条带有苯基异亮氨酸的侧链构成。然而,正是这种独特的构造,使得紫杉醇的合成之路充满了挑战。

研究人员对紫杉醇的生物合成路径进行了长达半个世纪的研究,但仍有许多关键的转化步骤隐藏在迷雾之中。特别是,氧化四环烷环的形成和C9位碳原子的氧化过程,至今仍是一个未解之谜,它们像两座高峰,阻挡在我们完全理解紫杉醇合成路径的道路上。
在这条合成路径中,找到负责氧化四环烷环和C9位氧化的关键酶,就像是找到了一把钥匙,能够将紫杉醇生物合成的各个孤立环节串联起来,形成一条完整的链条。
随着紫杉醇在癌症治疗中的应用越来越广泛,市场对它的需求也在不断攀升。然而,目前依赖天然资源提取的半合成生产方法,已经无法满足这种日益增长的需求。这就像一条狭窄的通道,无法承载日益增多的行人。
幸运的是,合成生物学的兴起为我们提供了一条新的道路。通过合成生物学,我们有望实现紫杉醇的绿色、可持续生产。因此,揭开紫杉醇生物合成路径的神秘面纱,不仅能够满足市场需求,更能够推动合成生物学的发展,开启一个全新的绿色制药时代。


环氧化催化关键酶-TOT1的解析
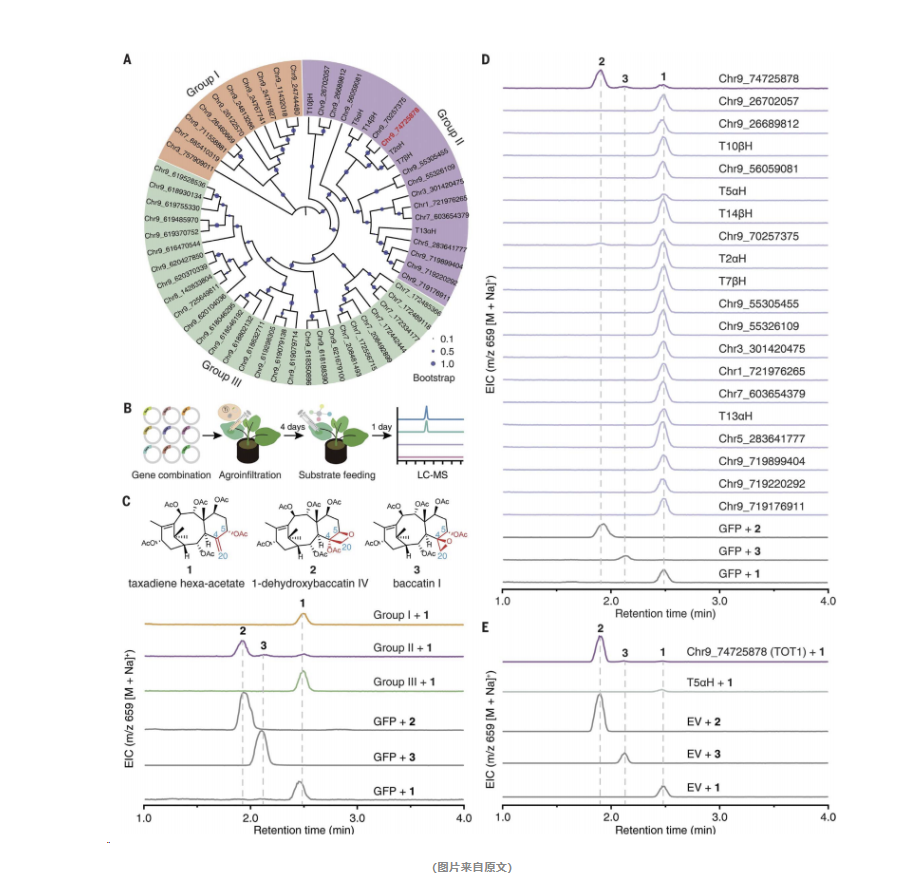
在探索紫杉醇这一神奇药物的背后,研究人员采取了一系列精妙的实验步骤,揭示了TOT1基因在氧化环形成中的关键角色。首先,他们运用了一种巧妙的筛选策略。基于紫杉烷生物合成途径的知识,研究人员将CYP725A家族的基因分为三个小组,并在烟草叶片中同时表达了这三组基因。随后,他们使用液相色谱质谱分析技术来检测底物的氧化产物。
接着,他们进行了活性筛选。在表达了第二组基因的叶片中,研究人员观察到了明显的底物氧化产物峰,这表明该组基因中可能含有他们寻找的酶。
为了验证这一假设,研究人员单独表达了第二组中的每一个基因。他们发现,当Chr9_74725878基因被表达时,底物氧化产物的数量显著增加。这一发现证明了该基因编码的蛋白质具有催化氧化环形成的能力。
基于这一发现,该基因被命名为TOT1,并通过体外表达和底物喂养实验进一步验证了其功能。
为了深入了解TOT1的作用机理,研究人员运用了结构模拟和反应机理计算的方法。他们的研究揭示了TOT1介导的氧化环形成反应机理,即它能直接将烯基转化为环氧化物和氧化环。
最后,为了验证TOT1在紫杉醇生物合成中的关键作用,研究人员在紫杉植物中敲低了TOT1的表达。他们观察到,百菌素和紫杉醇的含量随之降低,这一结果进一步证明了TOT1在紫杉醇生物合成中的重要性。
紫杉醇氧化烷基环形成机制
TOT1是一种具有双重功效的氧化酶。能够将紫杉醇中的烯基直接转化为环氧化物,进而形成氧化烷基环。这一过程无需经过环氧化物作为中间体的传统路径,而是通过一种更为直接和高效的转化方式。
在TOT1的催化下,烯基首先经历氧化反应,形成环氧化物。然而,这个环氧化物并非故事的终点,而是通过一系列结构重排,最终演变成为氧化烷基环。这一发现挑战了人们对氧化烷基环形成机制的传统认知,为理解紫杉醇的生物合成过程带来了新的视角。

通过精确的计算模拟和深入的结构分析,研究人员揭示了TOT1催化氧化烷基环形成的详细反应机理。他们观察到了过渡态和中间体的形成,这些都是在TOT1催化下发生的微妙变化。
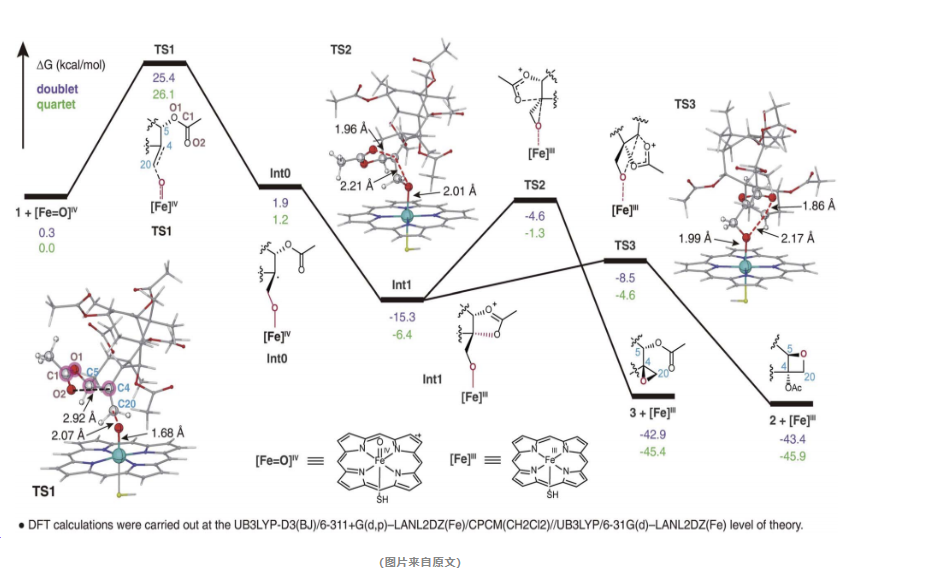
特别值得注意的是,模拟计算结果显示,氧化烷基环产物的形成在能量上和动力学上都是优先的,这与实验结果不谋而合。这一发现不仅验证了计算模型的准确性,也为紫杉醇的生物合成提供了新的科学依据。
这一机制的创新之处在于,它打破了我们以往对环氧化物作为中间体形成氧化烷基环的认知壁垒,为氧化烷基环的形成提供了全新的解释。
C9羟基化酶的突破
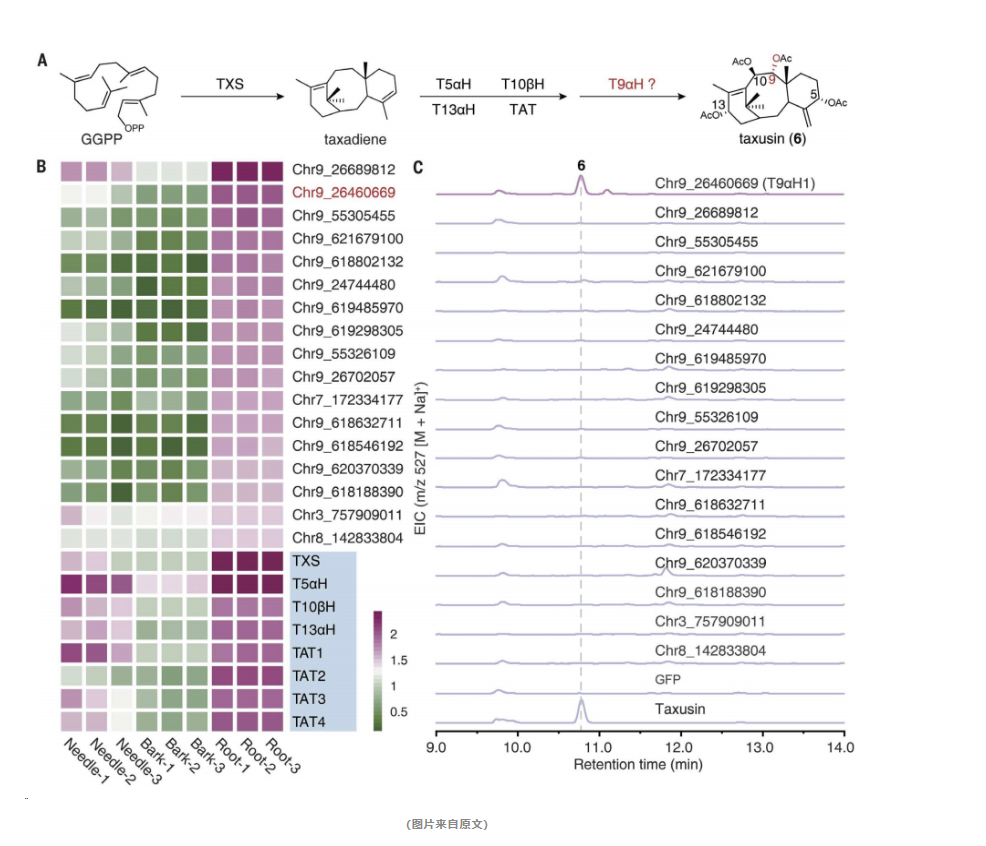
研究人员采取了一系列精确的实验步骤,最终揭示了关键的C9羟基化酶T9αH1。
首先,研究人员对不同组织中的紫衫素含量进行了分析,发现紫衫素主要在Taxus植物的根部积累,而在针叶和树皮中的含量则相对较低。
接着,他们对紫衫素生物合成途径中已知基因的表达模式进行了研究,发现这些基因在根部高度表达,与紫衫素的含量分布密切相关。
进一步地,研究人员对CYP725A家族基因的组织特异性表达模式进行了分析,并从中筛选出了17个潜在的候选基因。
为了鉴定这些候选基因中是否含有合成紫衫素所需的C9羟基化酶,研究人员在烟草植物中逐一表达了这些候选基因,并同时表达了已知的紫衫素生物合成基因。他们通过检测是否能够合成紫衫素来评估这些候选基因的功能。
实验结果显示,只有Chr9_26460669基因的表达使得烟草能够合成紫衫素。这一发现证实了Chr9_26460669基因编码了C9羟基化酶,并被命名为T9αH1。
为了进一步验证T9αH1的C9羟基化酶活性,研究人员在烟草中表达了T9αH1,并成功检测到了紫衫素的生成。
通过这一系列精妙的实验步骤,研究人员不仅成功从候选基因中筛选并鉴定出了C9羟基化酶T9αH1,而且填补了巴卡丁III生物合成途径中的一个关键空白。
人工重建巴卡丁III生物合成途径
研究人员首先在烟草叶片中同时共表达了已知参与巴卡丁III生物合成的基因,包括TXS、T5αH、T13αH、T2αH、T7βH、TAT和TBT。
接着,他们在上述已知基因的基础上,加入了新发现的两个关键基因TOT和T9αH,为巴卡丁III的生物合成途径增添了新的力量。
随后,研究人员在烟草叶片提取物中检测到了一个新产物峰,其质谱和保留时间与巴卡丁III完全一致,这表明他们可能已经成功合成巴卡丁III。
为了进一步确认这个新产物的身份,研究人员运用了MS/MS分析技术,结果确认该新产物确实是巴卡丁III。
为了鉴定巴卡丁III生物合成的核心途径,研究人员逐一移除了每个基因,发现只有当所有9个基因同时表达时才能产生巴卡丁III,这证明了这9个基因构成了巴卡丁III生物合成的核心途径。

此外,他们还发现TAT不仅可以催化C10位的乙酰化,而且T10βH在C10位氧化反应中可以被T5αH等其他酶取代。
特别值得注意的是,尽管DBAT被认为是C10位乙酰化的关键酶,但研究人员发现,即使在没有DBAT的情况下,巴卡丁III仍然可以合成,这表明TAT可能也具有C10位的乙酰化功能。
通过人工共表达关键基因,研究人员成功地在烟草中重建了巴卡丁III的生物合成途径,为进一步通过合成生物学生产紫杉醇提供了坚实的基础。
核心基因的功能协同
在探索巴卡丁 III 生物合成过程的奥秘中,研究人员发现了一系列核心基因之间功能协同性的显著特征。首先,TOT、T9αH等基因主要在根部表达,与下游基因的表达模式相似,表明这些基因在组织水平上存在协同表达的现象。
其次,经过茉莉酸处理后,这些基因的表达模式呈现协同上调的趋势,与下游基因的表达模式相似,这提示茉莉酸可能参与协同调控这些基因。
此外,这些基因的表达之间存在显著的正相关性,而与T14βH的表达模式不同,这表明它们之间存在协同调控关系。
在亚细胞水平上,这些酶主要定位于内质网,与质体和细胞质中的酶存在协同作用,形成协同代谢途径。
进一步地,这些基因构成的代谢网络呈现复杂的网络状而非线性,形成协同的代谢途径。
最后,这些基因在染色体上的分布位置存在关联,暗示它们之间可能存在协同进化关系。
综合来看,这些核心基因在表达模式、调控、亚细胞定位、代谢途径等方面存在协同性,共同参与巴卡丁 III 的生物合成过程。

综上所述,本研究确定了紫杉科植物中催化生成抗癌药物紫杉醇的关键酶。紫杉醇的结构复杂,包含一个功能化的二萜核心骨架(巴卡丁III)和苯异戊烯酰基侧链。尽管在过去五十年中进行了大量的研究,但巴卡丁III的完整生物合成途径仍不清楚。研究人员通过对紫杉醇生物合成途径的筛选,发现了一个双功能细胞色素P450酶TOT1,它催化了紫杉醇中氧杂环的形成。通过共表达其他已知的参与巴卡丁 III合成的基因,研究人员成功地在烟草中人工重建了巴卡丁 III的生物合成途径。这项研究为利用合成生物学手段绿色高效制造紫杉醇提供了可能。
翌圣助力产品
在该研究中,研究团队使用了翌圣生物染料法荧光定量qPCR试剂进行基因表达分析:

目前翌圣qPCR mix系列的产品已经荣登Nature、 Cell等多个顶级期刊,获得科研大牛们认可!以下仅展示部分助力发表的高分文章:
[1] Li Y, Wang D, Ping X, et al. Local hyperthermia therapy induces browning of white fat and treats obesity. Cell. 2022;185(6):949-966.e19.doi:10.1016/j.cell.2022.02.004.(IF=66.850)
[2] Seki T, Yang Y, Sun X, et al. Brown-fat-mediated tumour suppression by cold-altered global metabolism. Nature. 2022;608(7922):421-428. doi:10.1038/s41586-022-05030-3. (IF=69.504)
[3] Chen P, Wang W, Liu R, et al. Olfactory sensory experience regulates gliomagenesis via neuronal IGF1. Nature. 2022;606(7914):550-556. doi:10.1038/s41586-022-04719-9.(IF=69.504)
[4] Dong W, Zhu Y, Chang H, et al. An SHR-SCR module specifies legume cortical cell fate to enable nodulation. Nature. 2021;589(7843):586-590. doi:10.1038/s41586-020-3016-z.(IF=69.504)
[5] Lu XY, Shi XJ, Hu A, et al. Feeding induces cholesterol biosynthesis via the mTORC1-USP20-HMGCR axis. Nature. 2020;588(7838):479-484. doi:10.1038/s41586-020-2928-y.(IF=69.504)
[6] Bi X, Wang K, Yang L, et al. Tracing the genetic footprints of vertebrate landing in non-teleost ray-finned fishes. Cell. 2021;184(5):1377-1391.e14. doi:10.1016/j.cell.2021.01.046.(IF=66.850)
[7] Liu S, Hua Y, Wang J, et al. RNA polymerase III is required for the repair of DNA double-strand breaks by homologous recombination. Cell. 2021;184(5):1314-1329.e10. doi:10.1016/j.cell.2021.01.048.(IF=66.850)
[8] Liu CX, Li X, Nan F, et al. Structure and Degradation of Circular RNAs Regulate PKR Activation in Innate Immunity. Cell. 2019;177(4):865-880.e21. doi:10.1016/j.cell.2019.03.046.(IF=66.850)
[9] Han X, Wang R, Zhou Y, et al. Mapping the Mouse Cell Atlas by Microwell-Seq. Cell. 2018;172(5):1091-1107.e17. doi:10.1016/j.cell.2018.02.001.(IF=66.850)
[10] Chai Q, Yu S, Zhong Y, et al. A bacterial phospholipid phosphatase inhibits host pyroptosis by hijacking ubiquitin. Science. 2022;378(6616):eabq0132. doi:10.1126/science.abq0132.(IF=63.714)
[11] Yu Q, Liu S, Yu L, et al. RNA demethylation increases the yield and biomass of rice and potato plants in field trials. Nat Biotechnol. 2021;39(12):1581-1588. doi:10.1038/s41587-021-00982-9.(IF=68.164)
[12] Han F, Liu X, Chen C, et al. Hypercholesterolemia risk-associated GPR146 is an orphan G-protein coupled receptor that regulates blood cholesterol levels in humans and mice. Cell Res. 2020;30(4):363-365. doi:10.1038/s41422-020-0303-z.(IF=46.297)
[13] Wang Z, Lu Z, Lin S, et al. Leucine-tRNA-synthase-2-expressing B cells contribute to colorectal cancer immunoevasion. Immunity. 2022;55(6):1067-1081.e8. doi:10.1016/j.immuni.2022.04.017.(IF=43.474)
[14] Bi Q, Wang C, Cheng G, et al. Microglia-derived PDGFB promotes neuronal potassium currents to suppress basal sympathetic tonicity and limit hypertension. Immunity. 2022;55(8):1466-1482.e9. doi:10.1016/j.immuni.2022.06.018.(IF=43.474)
[15] Wang X, Ni L, Wan S, et al. Febrile Temperature Critically Controls the Differentiation and Pathogenicity of T Helper 17 Cells. Immunity. 2020;52(2):328-341.e5. doi:10.1016/j.immuni.2020.01.006.(IF=43.474)
[16] Xiao J, Li W, Zheng X, et al. Targeting 7-Dehydrocholesterol Reductase Integrates Cholesterol Metabolism and IRF3 Activation to Eliminate Infection. Immunity. 2020;52(1):109-122.e6. doi:10.1016/j.immuni.2019.11.015.(IF=43.474)
[17] Zhang X, Zhang C, Qiao M, et al. Depletion of BATF in CAR-T cells enhances antitumor activity by inducing resistance against exhaustion and formation of central memory cells. Cancer Cell. 2022;40(11):1407-1422.e7. doi:10.1016/j.ccell.2022.09.013.(IF=38.585)
[18] Wang XY, Wei Y, Hu B, et al. c-Myc-driven glycolysis polarizes functional regulatory B cells that trigger pathogenic inflammatory responses. Signal Transduct Target Ther. 2022;7(1):105. Published 2022 Apr 18. doi:10.1038/s41392-022-00948-6.(IF=38.104)
[19] Fan H, Hong B, Luo Y, et al. The effect of whey protein on viral infection and replication of SARS-CoV-2 and pangolin coronavirus in vitro. Signal Transduct Target Ther. 2020;5(1):275. Published 2020 Nov 24. doi:10.1038/s41392-020-00408-z.(IF=38.104)
[20] Ren Y, Wang A, Wu D, et al. Dual inhibition of innate immunity and apoptosis by human cytomegalovirus protein UL37x1 enables efficient virus replication. Nat Microbiol. 2022;7(7):1041-1053. doi:10.1038/s41564-022-01136-6.(IF=30.964)
<上下滑动查看更多>
翌圣生物基因研究完整解决方案

产品特别推荐
|
方法 |
分类 |
产品名称 |
货号 |
|
RNA提取 |
同Trizol提取 |
TRIeasy™ Total RNA Extraction Reagent |
|
|
免氯仿升级版 |
TRIeasy™ Total RNA Extraction Reagent(Tcm Free) |
||
|
动物组织/细胞总RNA提取,避开有毒试剂,最快15 min完成 |
MolPure® Cell/Tissue Total RNA Kit细胞/组织总RNA提取试剂盒 |
||
|
反转录试剂 |
5 min一步gDNA去除&反转录预混液(下游应用qPCR) |
Hifair® AdvanceFast One-step RT-gDNA Digestion SuperMix for qPCR |
|
|
5 min快速反转,最长可满足14 kb cDNA合成,含gDNA去除(下游应用PCR/qPCR) |
Hifair® AdvanceFast 1st Strand cDNA Synthesis Kit |
11149/11150ES |
|
|
高质量第一链cDNA合成预混液,含gDNA去除(下游应用qPCR) |
Hifair® III 1st Strand cDNA Synthesis SuperMix for qPCR (gDNA digester plus) |
||
|
qPCR染料法 |
高特异高荧光值定量预混液(染料法) |
Hieff UNICON® Advanced qPCR SYBR Master Mix |
|
|
高灵敏通用型定量预混液(染料法) |
Hieff UNICON® Universal Blue qPCR SYBR Master Mix |
||
|
超高性价比定量预混液 (染料法),已发文章累计IF达到5000+ |
Hieff® qPCR SYBR Green Master Mix (No Rox) |
||
|
Hieff® qPCR SYBR Green Master Mix (Low Rox) |
|||
|
Hieff® qPCR SYBR Green Master Mix (High Rox) |
